pISSN : 3058-423X eISSN: 3058-4302
Open Access, Peer-reviewed
pISSN : 3058-423X eISSN: 3058-4302
Open Access, Peer-reviewed
Jong Soo Choi,Byeong Su Kim,Yeon Woong Kim,Jin Hwa Choi,Dong Hoon Shin
http://dx.doi.org/10.17966/KJMM.2015.20.3.53 Epub 2015 October 07
Abstract
Background: Traditionally, mating types of dermatophytes had been identified by mating experiments.It took a long time and there were many limitations. Recently, we can figure out the fungal mating types using molecular mating
type analysis by detecting mating type (MAT) genes. The mating type (+) specific gene of the high-mobility-group (HMG) DNA binding domain and the mating type (-) specific gene of alpha-box were found in Arthroderma simii and A.
vanbreuseghemii.
Objective: We applied this molecular mating type analysis to strains of Trichophyton interdigitale, T. rubrum,
Microsporum canis in Korea and compared these results with previous reports.
Methods: Thirty-four strains of T. interdigitale (12 granular types, 9 powdery types, 8 purple-red types, 5 cottony types), 5 strains of T. rubrum, and 5 strains of M. canis were examined. We analyzed ribosomal RNA internal
transcribed space 1, 4 sequencing of T. interdigitale subtypes and investigated the mating type of dermatophytes
using alpha-box gene and HMG gene primers.
Results: Among 12 strains of granular type of T. interdigitale, 9 strains were type (-) and other 3 strains were type (+). All of them were zoophilic. All strains of powdery, purple-red and cottony types of T. interdigitale were type (+) and anthropophilic. In T. rubrum and M. canis, all strains were type (-). These results were matched with previously reported studies.
Conclusion: The molecular mating type analysis of dermatophytes was quicker method than conventional mating experiments. Moreover, MAT genes are highly conserved even in apparently asexual fungi. The results were well matched with previous reports with traditional mating tests.
Keywords
Dermatophytes Mating type gene Microsporum canis Trichophyton mentagrophytes Trichophyton rubrum
서 론
피부사상균은 피부와 부속기에 감염을 일으키는 진균으로 무성세대 (anamorph) 분류는 현미경으로 관찰하여 대분생자 (macroconidium)의 형태, 소분생자 (microconidium)의 발생 유무, 형태및 배열 등에 따라 Microsporum (M.), Trichophyton (T.), Epidermophyton (E.)의 3속 (genus)으로 분류한다. 완전세대 (유성세대, teleomorph)는 Arthroderma (A.) 속으로, 생태학적으로는 사람친화성 (anthropophilic), 동물친화성 (zoophilic), 흙친화성 (geophilic) 피부사상균으로 분류한다[1],[2]. 전 세계적으로 42종이 알려져 있고, 대한민국에서는 M. canis, M. ferrugineum, M. gypseum, M. nanum, T. rubrum, T. mentagrophytes, T. erinacei, T. verrucosum, T. violaceum, T. schoenleinii, T. tonsurans, E. floccosum 등 12종이 확인되었다[3],[4].
피부사상균의 완전세대형은 균종의 분류에 중요하며 교배시험 (mating test)을 이용하였다. 그러나 이 방법은 표준시험 균주와 4~6주의 배양 기간이 필요하고 성 능력을 잃어버린 균주는 교배형을 알 수 없고, T. rubrum, T. interdigitale와 같이 교배형 배우자를 잃어버린 경우 교배시험이 불가능하다는 단점이 있다[5].
최근에는 분자생물학적 방법으로 mating type (MAT) 유전자 분석을 통해 진균의 교배형을 빠르고 정확하게 알아낼 수 있다. Bubnick과 Smulian[6] 은 Ascomycetes에서 MAT 유전자를 발견하였으며 (+) 교배형은 MAT1-2의 HMG (High-mobility group) 유전자이고, (-) 교배형은 MAT1-1의 alphabox 유전자였다. Li 등[7]은 M. gypseum, M. canis, T. rubrum, T. tonsurans에서 MAT 유전자를 검출하였다. Kano 등[8],[9],[10]은 A. benhamiae, A. vanbreuseghemii, A. simii에서 HMG와 alpha-box 유전자를 발견하였고, T. interdigitale의 동물친화성 균주는 (+) 또는 (-) 교배형, 사람친화성 균주는 (+) 교배형임을 확인하였으며, T. rubrum은 모두 (-) 교배형임을 밝혔다.
T. mentagrophytes complex에는 A. vanbreuseghemi, A. simii, A. benhamiae의 3가지 완전세대형이 있다. A. vanbreuseghemi의 불완전세대형은 T. interdigitale이고 사람친화성 (T. mentagrophytes var. interdigitale)과 동물친화성이 있다[11]. 한국의 동물과 사람에서 분리되는 T. mentagrophytes complex는 대부분 T. interdigitale/A. vanbreuseghemii (T. mentagrophytes var. interdigitale)이며 형태적으로 과립형, 분말형, 도실형, 융모형으로 분류할 수 있고[12] 과립형은 동물친화성이며, 분말형, 도실형, 융모형은 사람 친화성으로 알려졌다[5]. Heidemann 등[13]은 ribosomal RNA (rRNA) internal transcribed spacer (ITS) 1, 5.8S, ITS2 염기서열 분석을 통해 T. interdigitale를 type I, II, III*, III, IV의 5가지 아형으로 분류하였고 type I과 II는 사람친화성, type III*, III, IV는 동물 친화성으로 구분하였다. Kim[14]은 한국인에서 분리된 T. interdigitale의 형태와 ITS 염기서열을 분석하여 분말형, 도실형, 융모형은 Heidemann 등의 type II와 동일하며 사람친화성이었고, 과립형은 Granular-a, Granular-b로 세분화하였다. Granular-a는 Heidemann 등의 type IV와 가까운 동물친화성이고 Granular-b는 Heidemann 등의 type II와 III* 의 중간에 위치하며 동물친화성과 사람친화성의 경계에 위치한다고 하였다.
본 연구에서는 MAT 유전자를 이용한 교배형 분석을 국내 환자로부터 분리된 T. interdigitale 아형, T. rubrum, M. canis 균주에 적용하여 그 정확도를 알아보고자 하였다.
재료 및 방법
1. 연구 대상
2011년 1월부터 2013년 12월까지 3년간 대구가톨릭피부과의원을 방문한 한국인 피부진균증 환자에서 분리된 T. interdigitale, T. rubrum, M. canis를 대상으로 하였다. 균주의 수는 T. interdigitale 34주 (과립형 12주, 분말형 9주, 도실형 8주, 융모형 5주), T. rubrum 5주, M. canis 5주이었다.
2. 배양 및 형태학적 균 동정
Cycloheximide (Sigma, St Louis, MO, USA) 0.5 mg/mL와 chloramphenicol (Sigma, St Louis, MO, USA) 0.05 mg/mL를 첨가한 Sabouraud's dextrose agar (SDA, Becton, Dickinson and Company, NJ, USA) 평판 배지에 접종하여 25℃에서 3주간 배양하였다.
배양하여 자란 균주의 집락 형태, 성장 속도 및 색깔 등의 육안적 소견과 lactophenol cotton blue로 염색하여 현미경으로 균사, 분생자 및 대분생자를 관찰하여 균을 동정하였다.
3. DNA 분리
DNA 분리를 위하여 QIAmp DNA Mini Kit (Qiagen, Hilden, Germany)를 사용하였다. SDA에 배양하여 순수 분리한 균 집락을 1.5 mL microcentrifuge tube에 넣고 animal tissue lysis (ATL) buffer 180 μL와 proteinase K 20 μL를 넣고 56℃에서 12시간 동안 처리하여 세포벽과 세포막을 분해하였다. 그 후 ATL buffer 200 μL를 넣고 섞은 후 70℃에서 10분간 가열하였다. 이 후 100% ethanol 200 μL를 넣고 원심분리한 후 상층액을 QIAamp Mini spin column에서 7,150 g로 1분간 원심분리하였다. AW1 buffer 500 μL와 AW2 buffer 500 μL으로 각각 세척한 후 AE buffer 50 μL으로 녹인 DNA를 -20℃에서 보관하며 실험에 사용하였다.
4. rRNA ITS 염기서열을 이용한 T. interdigitale의 동정
1) 시동체 (Primer)
T. interdigitale의 rRNA ITS 부위를 증폭하기 위해 마크로젠 (Seoul, Korea)에서 제작한 범진균시동체인 primer ITS1 (5'-TCCGTAGGTGAACCTGCGG-3')과 primer ITS4 (5'-TCCTCCGCTTATTGATATGC-3')를 사용하였다[15].
2) PCR 증폭
DNA polymerase (STD16-R500)와 dNTP, tracking dye (bromophenol blue), 젤 침강제 등이 섞여 분주되어 있는 Smart Taq Pre-Mix (Solgent, Daejeon, Korea) 0.2 mL PCR tube에 반응 혼합물 1 μL, smart Buffer 18 μL를 넣고 rRNA ITS 부위를 증폭하기 위하여 primer ITS1 1 μL, primer ITS4 1 μL를 넣어 PCR을 시행하였다. Thermal cycler는 Perkin Elmer 480 (Perkin Elmer, Norwalk, USA)을 사용하였다. 반응조건은 처음에 predenaturation (94℃, 5분) 후, denaturation (94℃, 30초), annealing (58℃, 30초), extension (72℃, 3분)을 30회 반복하였고 마지막에 final extension (72℃, 10분)을 하였다. PCR로 증폭된 DNA를 ethidium bromide가 첨가된 1% agarose gel에서 전기영동 (Mupid-2 Mini Gel Migration Trough, Cosmo Bio Co., LTD, Tokyo, Japan) 후 자외선 투과조명기로 관찰하였다.
3) 염기서열 분석
전기영동에서 증폭 산물이 확인된 PCR 산물을 purification kit (Bioneer, Daejeon, Korea)로 정제한 후 마크로젠에 의뢰하여 염기서열을 얻었고 GenBank의 BLAST 분석을 통해 분석하였다.
5. MAT 유전자를 이용한 교배형 분석
(+) 교배형의 HMG 유전자를 증폭하기 위해 primer TmHMG1S와 primer TmHMG1R을 사용하였고 (-) 교배형의 alpha-box 유전자를 증폭하기 위해 primer TmMATa1S와 primer TmMATa1R을 사용하였다[9].
2) PCR 증폭
Smart Taq Pre-Mix (Solgent) 0.2 mL PCR tube에 반응 혼합물 1 μL, smart Buffer 18 μL를 넣고 HMG gene, alpha-box gene 부위를 각각 증폭하기 위하여 primer TmHMG1S와 TmHMG1R 또는 TmMATa1S 와 TmMATa1R을 1 μL씩 두 개의 tube에 각각 넣어 PCR을 시행하였다. 반응은 94℃에서 5분 전 변성시킨 후, 94℃에서 1분, 55℃에서 1분, 72℃에서 2분의 조건으로 35회 반복하였고 72℃에서 10분 연장반응 시켰다[9]. 전기영동 후 자외선 투과 조명기로 증폭 산물을 관찰하였다.

3) 교배형 판독
HMG gene 시동체 (TmHMG1S, TmHMG1R)를 이용하여 524 bp의 DNA 증폭 산물이 확인될 경우 (+) 교배형, alpha-box gene 시동체 (TmMATa1S, TmMATa1R)를 이용하여 471 bp의 DNA 증폭 산물이 확인될 경우 (-) 교배형으로 판정하였다 (Fig. 1).
결 과
1. T. interdigitale 아형의 교배형
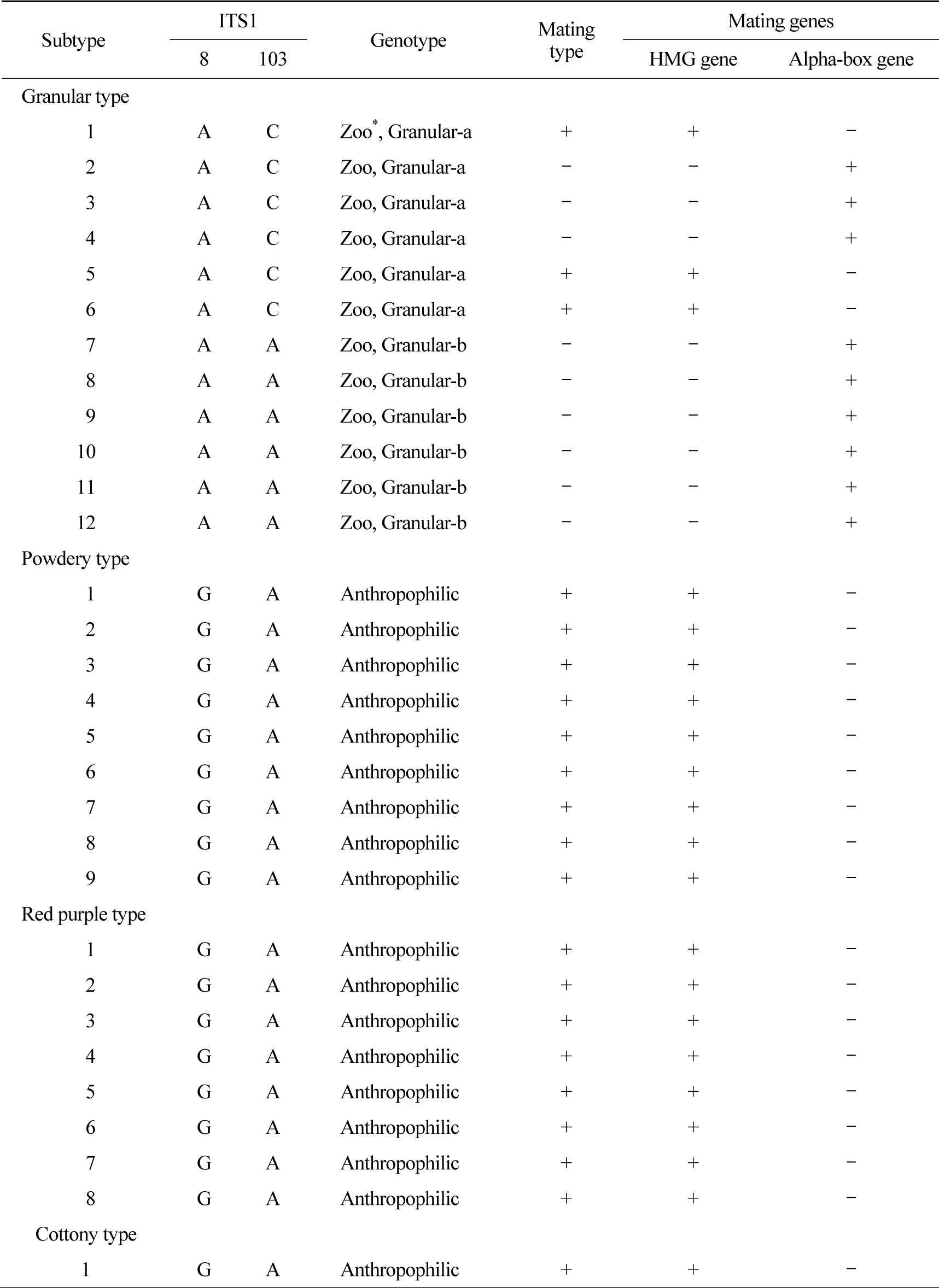
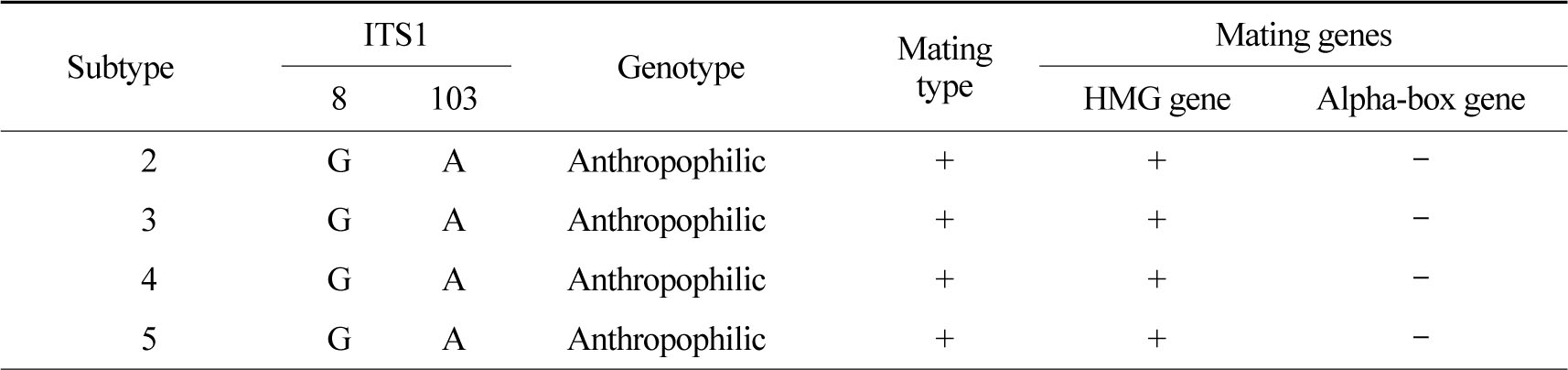
T. mentagrophytes의 ITS 염기서열 분석 결과 모든 균주가 T. interdigitale 이었다. 분말형, 도실형, 융모형은 모두 동일한 염기서열을 가지고 있었고, Heidemann 등[13]의 type II와 일치하여 사람친화성임을 알 수 있었다. 분말형 9주, 도실형 8주, 융모형 5주는 모두 (+) 교배형이었다 (Table 2).
T. interdigitale 과립형은 Granular-a 6주, Granularb 6주였다. 환자의 남녀 비는 Granular-a가 1:2, Granular-b는 2:1이었으며 평균 나이는 각각 42.8 ± 29.9세, 54 ± 17.8세이었다. Granular-a 환자는 병변의 위치가 얼굴 (5명), 다리 (1명) 순으로 많았고 Granular-b는 팔 2명, 가슴, 발바닥, 목, 얼굴각각 1명이었다. 병변이 발생한 부위를 부위 1
(팔, 다리, 몸통, 손, 목, 얼굴)과 부위 2 (발, 발톱, 사타구니, 엉덩이)로 구분하였을 때[14] Granular-a는 모두 (100%) 부위 1이었고 Granular-b는 5명 (83%)

이 부위 1, 1명 (17%)이 부위 2이었다. 과립형 12주 중 3주는 (+) 교배형, 9주는 (-) 교배형이었다. Granular-a는 (+) 교배형 3주, (-) 교배형 3주이었으며 Granular-b는 6주 모두 (-) 교배형이었다.
2. T. rubrum의 교배형
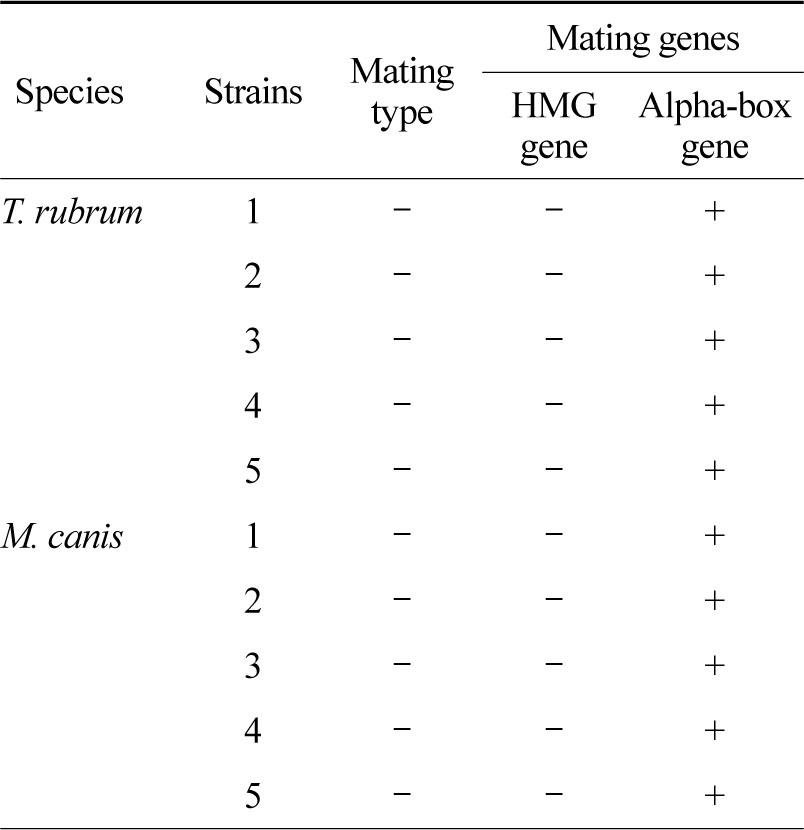
T. rubrum 5주는 HMG gene (-), Alpha-box gene (+)으로 모든 균주에서 (-) 교배형이었다 (Table 3).
*Zoo: zoophilic
3. M. canis의 교배형
M. canis 5주는 HMG gene (-), Alpha-box gene (+)으로 모든 균주에서 (-) 교배형이었다 (Table 3).
고 찰
본 연구에서 사용한 시동체들은 T. mentagrophytes의 염기서열을 바탕으로 제작되었다[9]. HMG 유전자와 alpha-box 유전자의 염기서열이 피부사상균 균종간에 다소 차이가 있으나, 공통부위에서 시동체를 제작하여 여러 피부사상균에서 사용이 가능하였다.
Suh와 Kim[5]은 T. mentagrophytes를 과립형, 분말형, 융모형으로 분류하여 교배시험을 하였다. 과립형 240주 중 109주 (45%)가 (+) 교배형, 131주 (55%)가 (-) 교배형이었다. 분말형 66주 중 53주(80%)가 (+) 교배형, 13주 (20%)가 (-) 교배형이었다. 융모형은 7주 중 5주 (71%)가 (+) 교배형, 2주 (29%)가 (-) 교배형이었다. Bang과 Lee[16]는 T. mentagrophytes를 과립형, 분말형으로 분류하여 교배시험을 하였으며 과립형 261주 중 42주 (16%)가 (+) 교배형, 219주 (84%)가 (-) 교배형이었다. 분말형은 218주 중 207주 (95%)가 (+) 교배형, 11주 (5%)가 (-) 교배형이었다. Kano 등[9]은 T. interdigitale를 동물친화성 균주와 사람친화성 균주로 분류하여 MAT 유전자 PCR을 이용해서 교배형을 분석하였고 동물친화성 15주 중 10주 (67%)가 (+) 교배형, 5주 (33%)가 (-) 교배형이었다.
사람친화성 72주는 모두 (100%) (+) 교배형으로 나타났다. 본 연구에서는 과립형 12주 중 (+) 교배형 3주 (25%), (-) 교배형 9주 (75%)이었고, 분말형 9주, 도실형 8주, 융모형 5주 모두 (100%) (+) 교배형으로 나타났다 (Table 4). 국내에서 시행한 교배시험과 본 연구에서는 동물친화성 균주에서 (-) 교배형이 더 많은 비율을 차지하였지만 일본에서 시행한 MAT 유전자 검출에서는 (+) 교배형이 더 많았다[9]. 이는 본 연구에 사용한 균주중에서 Granular-b형이 많았기 때문으로 생각되며 앞으로 더 많은 균주를 대상으로 실험하여 결과를 비교하여야 한다.
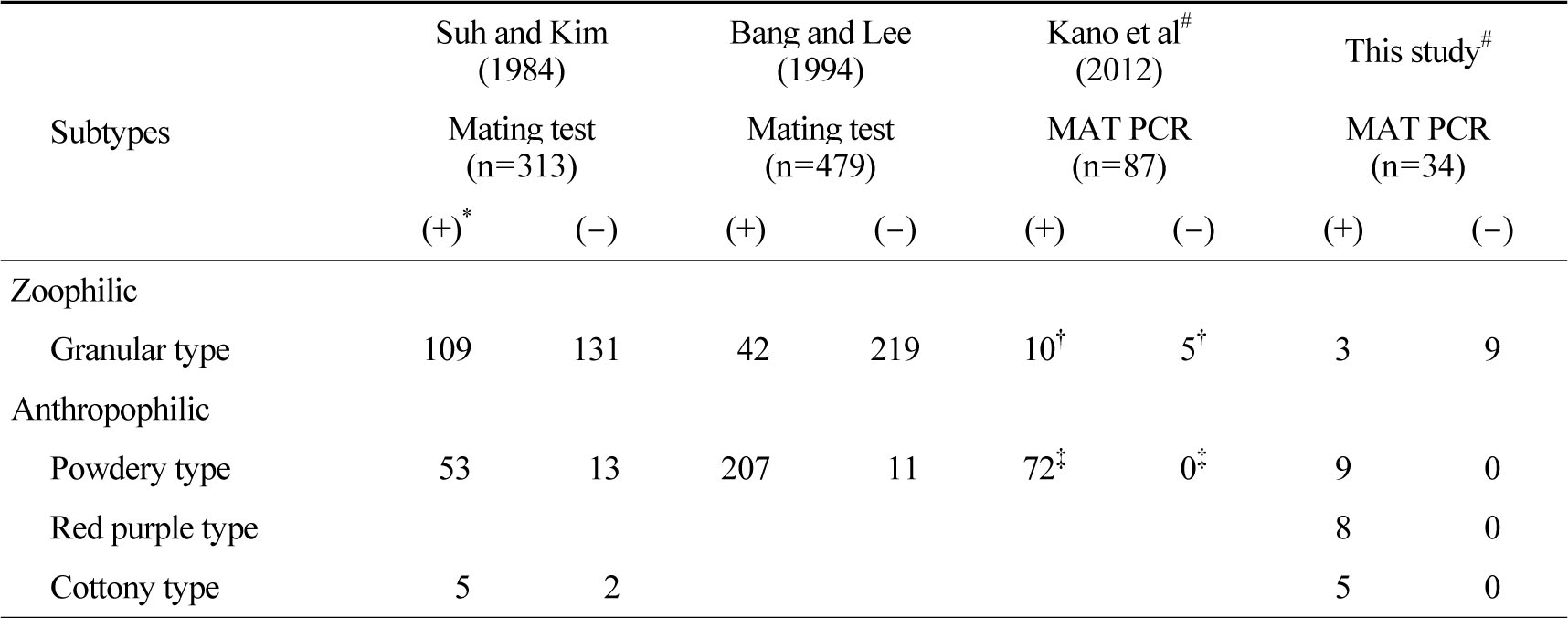
MAT: mating type genes
*(+): (+) mating type; (-): (-) mating type, †zoophilic, ‡anthropophilic
#T. interdigitale confirmed with ITS sequencing
사람친화성 T. mentagrophytes 는 분말형, 도실형, 융모형이 속하고, 모두 (+) 교배형이며 (-) 교배형은 알려져 있지 않으며, 동물친화성 T. mentagrophytes에는 과립형이 속하며 (+)교배형 또는 (-) 교배형이다[9],[14]. 본 연구에서 분말형, 도실형, 융모형이 모두 (+) 교배형이고, 과립형은 (-) 교배형이 많은 것도 각각 사람친화성, 동물친화성임을 의미한다. Georg1는 T. mentagrophytes의 과립형과 분말형이 상호 변환할 수 있고, 두 아형은 동일한 균종이라고 하였다. 그러나 과립형이 pleomorphism으로 인하여 분말형으로 변하더라도 ITS 염기서열은 변하지 않는다. 따라서 전통적 교배시험에서 분말형, 도실형, 융모형 균주가 (-) 교배형이 나온 것은, 과립형이 분말형, 융모형으로 변했을 가능성이 있다.
사람에서 검출된 T. mentagrophytes의 HMG 유전자는 A. vanbreuseghemii (+) 교배형의 HMG 유전자와 100% 일치하였다[9]. 따라서 사람친화성 T. mentagrophytes (T. interdigitale)는 A. vanbreuseghemii(+) 교배형으로부터 유래하였고, 진화과정에서 교배능력과 교배 배우자를 잃어버렸을 것으로 추정하였다.
Heidemann 등[13]은 T. interdigitale의 ITS 염기서열 분석을 통해 5가지 아형으로 분류하였으며 염기서열 8번과 103번이 각각 G와 A인 TI type I, II는 사람친화성, A와 C인 TI type III*, III, IV는 동물친화성이라 하였다. Kim[14]은 T. mentagrophytes의 분말형, 도실형, 융모형의 ITS 염기서열을 분석하여 형태와 상관없이 염기서열이 동일하였으며, Heidemann 등[13]의 TI type II (T. interdigitale typeII, GenBank accession number AF506036)와 100% 일치하였고 모두 사람친화성이었다고 보고하였다. 과립형 중 Granular-a는 ITS 염기서열 8번과 103번이 각각 A와 C로 동물친화성임을 밝혀내었다. Granular-b는 각각 A와 A로 사람친화성과 동물친화성의 중간이라고 하였다 (Table 5). 본 연구에서 Granular-b는 팔, 다리, 몸통, 손, 목, 얼굴에서만 분리되었으며, MAT 유전자 분석 결과 6주 모두 (-) 교배형이었다. 따라서 Granular-b형은 사람친화성은 아니며 동물친화성일 것이라 추측할 수 있었다. 하지만 균주수가 적으므로 추가적인 연구가 필요하다.
Weitzman과 Padhye[17]는 M. canis 198균주에서 교배시험을 하였으며 모두 (-) 교배형이었다. Suh등[18]은 M. canis 134주를 대상으로 교배시험을 하였으며 112주 (84%)에서 (-) 교배형, 22주 (16%)는 반응하지 않았고, (+) 교배형은 없었다. 본 연구에서도 M. canis 5주 모두 (-) 교배형이었다.
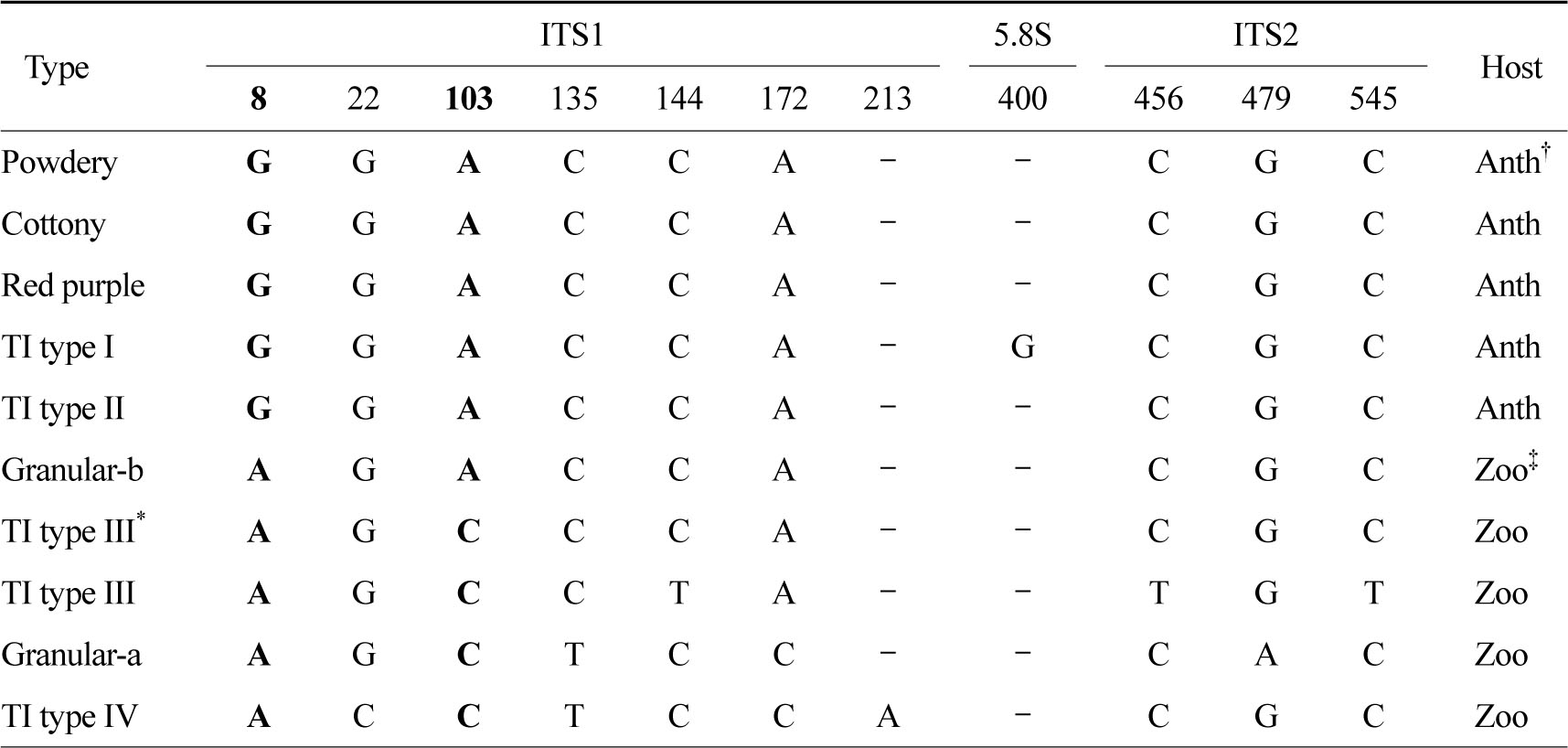
†Anth: anthropophilic; ‡Zoo: zoophilic
Signature polymorphism of T. interdigitale
Ranganathan 등[19]의 T. rubrum 교배시험에서 25개 균주 모두 (-) 교배형이었다. Kano 등[10]이 일본에서 분리된 T. rubrum 216주에서 MAT 유전자 PCR을 시행하였으며 모든 균주에서 alpha-box 유전자 양성, 즉 (-) 교배형이었다.본 연구에서도 T. rubrum 5주 모두 (-) 교배형이었다.
본 연구에서는 MAT 유전자를 이용하여 T. interdigitale, M. canis, T. rubrum의 교배형 분석을 시행하였고, 교배시험을 이용한 기존의 보고와 일치함을 알 수 있었다. 동일한 균주를 이용하여 MAT 유전자 분석과 교배시험을 동시에 시행하여 그 결과를 직접 비교하지 못한 점과 개체수가 적어 통계적 유의성을 비교하지 못한 점 등이 제한점으로 생각된다.
MAT 유전자 분석은 교배시험과 비교하여 짧은 시간에 시행할 수 있었으며 진화 과정에서 교배 능력이나 교배 배우자를 잃어버린 경우를 포함한 모든 균주에서 교배형을 알아낼 수 있었다. 따라서 향후 계통발생학적 연구 또는 유성세대 연구에서 기존의 교배시험을 대체하여 MAT 유전자 분석이 유용하게 사용될 수 있을 것이다.
결 론
피부사상균의 교배형을 알기 위해 교배시험이 널리 사용되어 왔다. 이 방법은 시간이 많이 걸리는 등 많은 제한점이 있었다. 최근에는 분자생 물학적인 방법으로 MAT 유전자를 검출하여 진균의 교배형을 알아낼 수 있다. (+) 교배형은 HMG 유전자, (-) 교배형은 alpha-box 유전자를 각각 검출할 수 있다. 본 연구에서는 MAT 유전자를 이용한 교배형 분석을 국내의 T. interdigitale 아형, T. rubrum, M. canis 균주에 적용하여 교배시험을 이용한 결과와 비교하였다. 2011년 1월부터 2013년 12월까지 대구가톨릭피부과의원을 방문한 한국인 피부진균증 환자에서 분리된 T. interdigitale 34주 (과립형 12주, 분말형 9주, 도실형 8주, 융모형 5주), T. rubrum 5주, M. canis 5주를 대상으로 하였다. PCR을 이용하여 T. interdigitale의 ribosomal RNA internal transcribed space 염기서열 분석을 하였고 HMG 유전자 시동체와 alpha-box 유전자 시동체를 이용하여 모든 균주의 MAT 유전자 증폭 산물을 확인하였다. T. interdigitale 과립형 12주 중 3주는 (+) 교배형, 9주는 (-) 교배형이었으며 모두 동물친화성이었다. T. interdigitale 분말형, 도실형, 융모형은 사람친화성이며 모두 (+) 교배형이었다. T. rubrum과 M. canis는 모두 (-) 교배형이었다. 이들은 모두 문헌에 보고된 교배시험의 결과와 일치하였다. 분자생물학적 방법을 이용한 피부사상균 교배형 분석은 짧은 시간에 시행할 수 있었고 모든 균주에서 결과를 알 수 있었으며, 기존에 보고된 교배시험 결과와 일치하였다.
Conflict of interest
The authors declare that there are no conflicts of interest.
References
1. Georg LK. The relationship between the downy and granular forms of Trichophyton mentagrophytes. J Invest Dermatol 1954;23:123-141
Google Scholar
2. Ajello L. Present day concepts of the dermatophytes. Mycopathologia 1962;17:315-324
Google Scholar
3. Kim KH. Changing patterns of dermatophytosis and its causative agents according to social and economic developments in Korea. Korean J Med Mycol 2006; 11:1-12
Google Scholar
4. Rhee DY, Kim MS, Chang SE, Lee MW, Choi JH, Moon KC, et al. A case of tinea manuum caused by Trichophyton mentagrophytes var. erinacei: the first isolation in Korea. Mycoses 2009;52:287-290
Google Scholar
5. Suh SB, Kim SW. Perfect state of Trichophyton mentagrophytes and their related infections. Korean J Dermatol 1984;22:610-618
Google Scholar
6. Bubnick M, Smulian AG. The MAT1 locus of Histoplasma capsulatum is responsive in a mating typespecific manner. Eukaryot Cell 2007;6:616-621
Google Scholar
7. Li W, Metin B, White TC, Heitman J. Organization and evolutionary trajectory of the mating type (MAT) locus in dermatophyte and dimorphic fungal pathogens. Eukaryot Cell 2010;9:46-58
Google Scholar
8. Kano R, Yamada T, Makimura K, Kawasaki M, Mochizuki T, Kamata H, et al. Arthroderma benhamiae (the teleomorph of Trichophyton mentagrophytes) mating type-specific genes. Mycopathologia 2011;171:333-337
Google Scholar
9. Kano R, Kawasaki M, Mochizuki T, Hiruma M, Hasegawa A. Mating genes of the Trichophyton mentagrophytes complex. Mycopathologia 2012;173: 103-112
Google Scholar
10. Kano R, Isizuka M, Hiruma M, Mochizuki T, Kamata H, Hasegawa A. Mating type gene (MAT1-1) in Japanese isolates of Trichophyton rubrum. Mycopathologia 2013;175:171-173
Google Scholar
11. Nenoff P, Herrmann J, Graser Y. Trichophyton mentagrophytes sive interdigitale? A dermatophyte in the course of time. J Dtsch Dermatol Ges 2007;5:198 -202
Google Scholar
12. Kim KH, Moon BC, Choi JS. Subtypes and mycologic characteristics of Trichophyton genus isolated in Taegu Korea. Korean J Med Mycol 1997;2:129-143
Google Scholar
13. Heidemann S, Monod M, Graser Y. Signature polymorphisms in the internal transcribed spacer region relevant for the differentiation of zoophilic and anthropophilic strains of Trichophyton interdigitale and other species of T. mentagrophytes sensu lato. Br J Dermatol 2010;162:282-295
Google Scholar
14. Kim WJ. Classification and typing of Trichophyton mentagrophytes isolated from Korean. Department of dermatology, graduate school, Yeungnam university 2012:1-27
15. White TJ, Bruns T, Lee S, Taylor JW. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. In: Innis MA, Gelfand DH, Sninsky JJ, White TJ, 'editors'. PCR protocols: a guide to mehods and applications. New York, N.Y: Academic Press, Inc., 1990:315-322
16. Bang YJ, Lee KS. Mating type and its mycological characteristics of Trichophyton mentagrophytes. Ann Lab Med 1994;26:84-89
17. Weitzman I, Padhye AA. Mating behaviour of Nannizzia otae (=Microsporum canis). Mycopathologia 1978;64:17-22
Google Scholar
18. Suh SB, Kim DW, Jun JB. Mating study of Microsporum canis isolated in Korea. Korean J Dermatol 1983;21:557-561
Google Scholar
19. Ranganathan S, Balajee SA, Menon T. Mating patterns of dermatophytes of diverse origin in India. Mycopathologia 1996;136:91-94
Google Scholar