pISSN : 3058-423X eISSN: 3058-4302
Open Access, Peer-reviewed
pISSN : 3058-423X eISSN: 3058-4302
Open Access, Peer-reviewed
Joon Goon Kim,Dong Hoon Shin,Jong Soo Choi,Chae Hoon Lee
http://dx.doi.org/10.17966/KJMM.2017.22.2.62 Epub 2017 June 25
Abstract
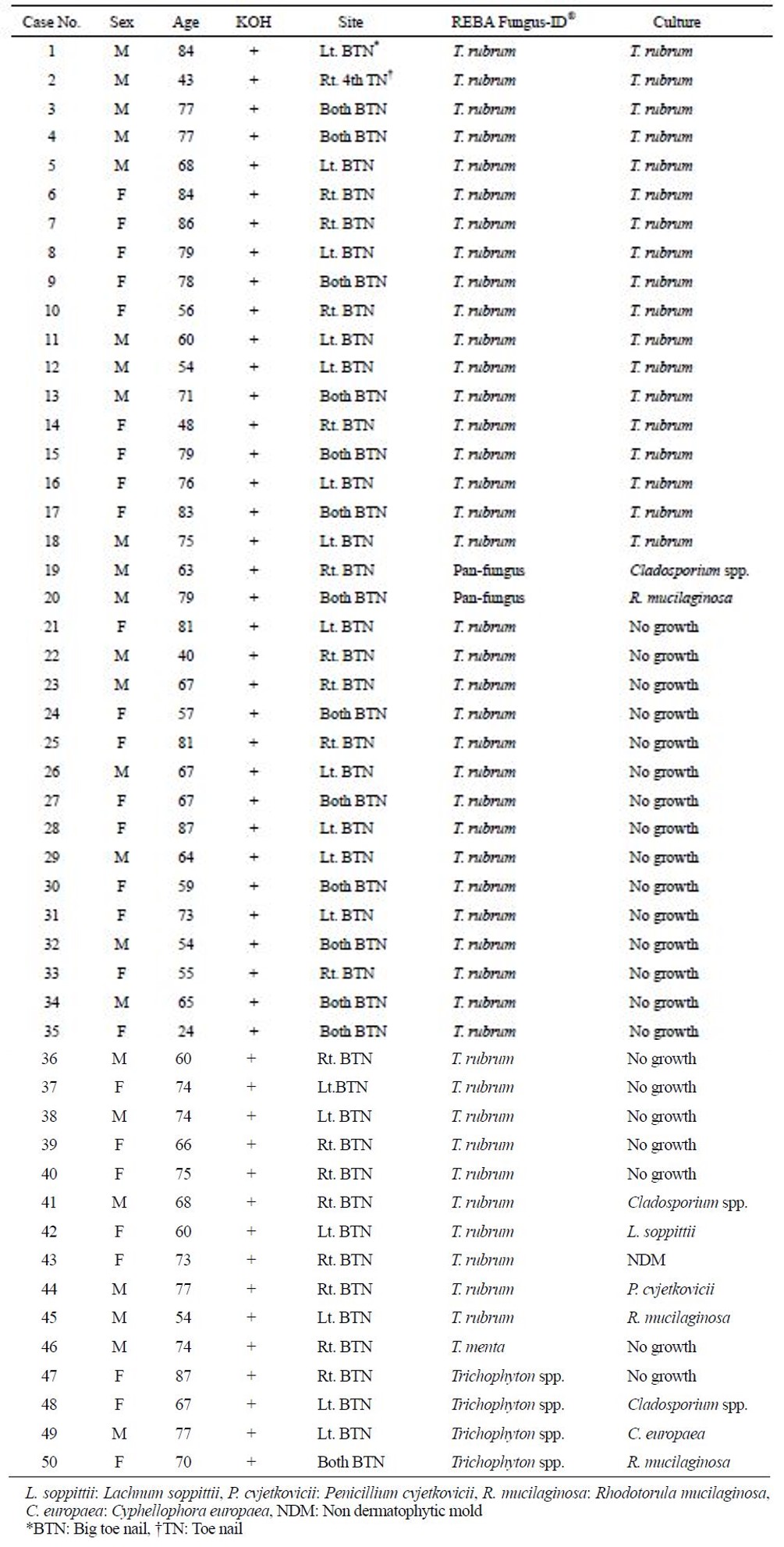
Objective: This study aims to compare the diagnostic efficacy of fungal culture and REBA Fungus- ID® which is a commercial PCR-REBA-based kit used for onychomycosis diagnosis.
Methods: Fifty nail samples were collected from 50 patients diagnosed with onychomycosis via direct microscopic examination using KOH preparation, and subjected to fungal culture and REBA Fungus-ID® test.
Results: The sensitivity of conventional fungal culture and REBA Fungus-ID® was 56% and 100%, respectively. In REBA Fungus-ID®, 43 of 50 samples were found to be infected with Trichophyton rubrum. Four of the remaining 7 samples were identified as infected with Trichophyton spp., one with Trichophyton mentagrophytes, and two revealed a panfungal DNA sequence. In fungal culture, 28 of 50 samples showed growth, of which 18 samples were identified as T. rubrum, 3 as Rhodotorula mucilaginosa, 3 as Cladosporium spp., 1 as Cyphellophora europaea, 1 as Penicillium cvjetkovicii, 1 as Lachnum soppittii, and 1 as non-dermatophytic mold. REBA Fungus-ID® and fungal culture were identical in 20 cases (40%). The non-dermatophytic fungi identified in fungal culture were considered contaminants.
Conclusion: Nail specimens can be used directly for REBA Fungus-ID®, which has a high sensitivity for onychomycosis diagnosis. Therefore, it can be considered useful for diagnosis and identification of the causative organism in mixed infections like onychomycosis.
Keywords
Molecular based method Onychomycosis PCR-REBA Polymerase chain reaction Reverse blot hybridization assay
서 론
손발톱진균증은 손발톱질환의 약 50% 정도를 차지하며, 임상에서 건선, 편평태선, 외상에 의한 손발톱변형과 같은 손발톱질환과 혼동될 수 있다[2]. 균종에 따라 항진균제 감수성이 다르므로 적절한 항진균제 선택을 위해 원인균의 동정이 반드시 필요하다[3].
손발톱진균증의 진단을 위한 표준 진단법은 KOH 검사로 진균을 관찰하고 진균배양을 통해 균종을 동정하는 것이다. 직접 균을 관찰하는 것은 진균감염을 빠르게 확인할 수 있으나 균종은 알 수 없다. 진균배양은 균의 형태학적 정보를 제공함으로써 균종을 알 수 있으나, 민감도(sensi- tivity)가 낮고 위음성(false negative)의 가능성이 높다[4]. 진균이 자라는 데까지 3주에서 4주 가량 시간이 많이 소요되고, 때로는 정확한 원인균의 확인을 위해 추가적인 계대배양이 필요하며, 균종의 특징적인 형태를 보이지 않는 경우도 있다[5].
최근에는 정확하고 빠르게 진균을 확인하기 위해서 다양한 분자생물학적 방법이 개발되고 있으며, polymerase chain reaction (PCR), PCR-enzyme linked immunosorbent assay (ELISA), restriction frag- ment length pattern (RFLP) analysis, matrix-assisted laser desorption-ionization-time of flight mass spectrometry (MALDI-TOF MS), real-time PCR 등이 있다[6]. 특히 진균의 리보솜 DNA 유전자 복합체의 internal transcribed spacer (ITS) 부위는 다양한 진균의 종을 확인하기 위해 사용돼 왔다[7]. 분자생물학적 진단방법은 진균을 확인하고 진단해내는데 있어서 고식적 방법과 비교했을 때 높은 민감도와 특이도를 가진다[8]. 하지만 진단과정에 있어 숙련자가 필요하고, 혼합감염이 된 경우에는 검체 직접 사용이 힘들 수 있고, 비싼 기기와 진단 시약이 사용되기 때문에 손발톱진균증과 같은 치명적이지 않은 질환에서 이러한 방법의 사용은 비용 대비 효율성에서 논란이 되어 왔다[9].
PCR-reverse blot hybridization assay (PCR-REBA)는 결과를 빠르게 알 수 있고 민감도와 특이도가 높다. 또한 검체를 직접 사용 가능해 진균배양에 소요되는 시간을 단축할 수 있으며, 상대적으로 다른 PCR 기반 진단방법보다 가격이 저렴하므로 손발톱진균증의 진단에서 장점을 가질 수 있다[10],[11],[12]. PCR-REBA는 종 특이 primer와 probe를 가지고 있기 때문에 위양성률이 낮으며, 한 번에 여러 개의 진균을 확인할 수 있고, 필요에 따라 임상적으로 중요한 진균의 probe들을 모아 검사할 수 있으므로 임상검체에 활용이 더욱 용이하다[13],[14].
REBA Fungus-ID® (M&D, Wonju, Korea)는 대표적인 의진균 6속(genus) 15종(species)의 동정이 가능한 분자 진단 키트로서,
PCR-REBA의 원리를 이용한다. PCR 증폭산물을 probe가 부착되어 있는 얇은 막에 결합시킨 후, 발색반응 결과물을 관찰하는 원리를 사용하고, 배양균, 손톱, 발톱 등 다양한 검체로부터 의진균의
검출 및 동정이 가능하다. 이에 본 연구자는 손발톱진균증에서 원인 균주 확인을 위해 고식적으로 시행되던 진균배양 검사에 비해서 분자생물학적 진단방법인 REBA Fungus-ID®의 유용성 여부를 확인하고자
실험을 진행하였다.
재료 및 방법
1. 연구 대상
2016년 5월부터 2016년 10월까지 영남대학교 병원에 입원한 환자 중 손발톱의 비후, 변색, 박리 등의 소견이 관찰되어 임상적으로 손발톱진균증이 의심되는 환자의 검체를 채취하여 KOH 검사를 시행하였으며, 균사가 관찰된 50명을 대상으로 하였다.
2. 실험방법
각각의 손발톱은 소독된 손톱깎이로 병변의 근위부까지 잘라낸 후 진균배양 검사와 REBA Fungus-ID®를 시행하는 데 사용하였다.
1) 진균배양 검사
(1) Potato corn meal tween
80 agar 배지 제작
증류수 500 ml에
potato dextrose agar (MB cell, USA) 10 g, corn meal agar (Difco Lab., USA) 10
g, peptone
agar (DUKSAN, Korea) 1 g을 섞고 중탕한 뒤 tween 80 (Showa, Japan) 3 ml를 넣어 멸균 소독한 후
chloramphenicol 250 mg과 cycloheximide
250 mg을 첨가하고 멸균된 페트리디쉬에 4 mm 두께로
분주하여 배지를 만들어
굳힌 다음 냉장고에 보관하였으며, 각 배지는 제작 후 1개월 이내에
사용하였다.
(2) 진균배양
각각의 검체를 평판 배지의 3부위에 접종하였으며, 25℃에서 2주에서 4주간 배양하였다.
(3) 진균동정
전형적인 집락과 현미경
소견 상 균종 특이적인 소견을 보고 동정을 하였으며, 균종 규명이 어려운 경우에는 rDNA
ITS 염기서열로 동정하였다. 집락을 채취하여 DNA 추출 후 마크로젠(Seoul,
Korea)에서 제작한 범진균 primer인 primer ITS-1 (5'-TCCGTAGGTGAACCTGCGG-3')과 primer ITS4 (5'-TCCTCCGCTTATTGATATGC-3')을 사용하여 PCR 시행하였으며 이 산물을 마크로젠에 의뢰하여 염기서열을 얻었다. 이후 최종적으로 GenBank에서 일치하는 균주를 검색하였다.
2) REBA Fungus-ID®
(1) DNA 분리
QIAmp DNA Mini Kit (Qiagen, Hilden, Germany)를 사용하였다. 손발톱 절편 3 mm 정도를 1.5 ml microcentrifuge tube에 넣고 animal tissue lysis (ATL) buffer 180 μl와 proteinase K 20 μl를 넣고 56℃에서 24시간 동안 처리하여 발톱 기질, 진균의 세포벽과 세포막을 분해하였다. 이후 ATL buffer 200 μl를 넣고 원심분리하였으며 상층액을 QIAmp Mini spin column에서 8,000 rpm으로 1분간 원심분리 하였다. AW1 buffer 500 μl와 AW2 buffer 500 μl으로 각각 세척한 후 AE buffer 50 μl으로 녹여 DNA를 얻었다.
(2) PCR
추출한 DNA를 이용하여 ITS-1 부위를 표적으로 한 one-tube nested PCR을 시행하였다. PCR을 위한 조성은 2 X PCR premix 10 μl,
primer mix 5 μl, 증류수 2 μl, 검체 DNA 3 μl이었다. PCR
반응은
초기변성을 위해 94℃ 5분, 1차 증폭을 위해 94℃ 30초, 60℃ 30초 반응을 10 cycles, 2차 증폭을 위해 94℃ 30초, 54℃ 30초 반응을 40 cycles 시행하였으며, 완전한 신장반응을 위해 72℃로 7분간 반응시켰다.
(3) REBA
앞의 반응 산물과 변성 용액(0.2 N NaOH, 0.2 mM EDTA)을 동량 혼합하여 실온에서 5분간 반응시켜 PCR 반응 산물을 단일 가닥으로 변성하였다. 이후 블로팅 트레이(blotting tray)의 홈(well)에 균 특이 올리고 nucleotide probe가 부착된 얇은 막(Fig. 1), 반응 산물, 900 μl의 교잡 용액(2 X SSPE/0.1% SDS)을 넣은 후 변성된 단일 가닥의 PCR 산물이 얇은 막에 부착된 probe와 반응하도록 55℃에서 30분간 교반하였다. 흡입기로 반응 산물을 완전히 제거하고, 얇은 막을 미리 데워둔 세척 용액(2 X SSPE/0.5% SDS)으로 55℃에서 10분간 교반하여 씻어내는 것을 2회 반복하여 비특이 산물을 세척하였다. 세척 용액을 완전히 제거한 뒤 미리 준비해 놓은 conjugate dilution solution (CDS)로 1:2,000배 희석된 streptavidin-alkaline phosphatase (AP) conjugate를 얇은 막 전체에 도포한 뒤 잘 반응하도록 30분간 상온에서 교반하였다. 이후 AP conjugate를 제거하고 다시 1 ml CDS 용액으로 상온에서 2회 세척한 후 발색반응을 위해 1:50으로 희석된 NBT/BCIP 용액을 첨가하였으며, 5분에서 10분간 발색반응을 기다린 후 증류수로 씻어내고 최종적으로 결과를 확인하였다.
(4) 결과판독
검사 결과는 Fig. 2과 같이 REBA Fungus-ID®의 매뉴얼대로 해석하였다. Pan-fungus와 Trichophyton (T.) species (spp.) 부위에 발색반응을 보인 경우에는 Trichophyton spp.로 판독하였으며, pan-fungus에만 발색반응을 보인 경우에는 REBA Fungus-ID®로 동정 가능한 6속(genus) 15종(species)에 속하지 않는 진균으로 판단하였고, 이를 pan-fungus로 기술하였다.
결 과
1. 검사 민감도(Sensitivity) 비교
임상 양상 및 KOH 검사 상에서 균사가 확인되어 손발톱진균증으로 확진된 환자를 대상으로 실험을 진행하여 진균배양 검사와 REBA Fungus-ID® 간의 검사 민감도를 비교할 수 있었다. 진균배양 검사에서는 총 50예 중 28예에서 양성을 보여 56%, REBA Fungus-ID®에서는 총 50예 중 50예에서 양성을 보여 100%의 민감도를 나타내어 REBA Fungus-ID®가 진균배양 검사에 비해 높은 민감도를 나타내었다(Table 1).
2. 진균배양 검사 소견
50예 중 28예에서 진균이 배양되었다. 배양된 28예 중 18예(64%)에서 T. rubrum이 동정되었다. 이 중 15예는 붉은 포도주색의 집락과 전형적인 현미경 소견을 보였으며, 3예는 rDNA ITS 염기서열분석을 통해 동정되었다. 나머지 10예는 Clado- sporium (C.) spp. 3예, Rhodotorula (R.) mucilaginosa 3예, Cyphellophora (C.) europaea 1예, Lachnum (L.) soppittii 1예, Penicillium (P.) cvjetkovicii 1예였다.
3. REBA Fungus-ID® 소견
50예 중 50예 모두에서 양성반응을 보였다. 그 중 43예(86%)에서 T. rubrum이 동정되었고, 1예는 T. mentagrophytes, 4예는 기타 Trichophyton spp., 2예는 pan-fungus로 동정되었다.
4. 진균배양 검사와 REBA Fungus-ID® 간의 일치 정도(Table 2)
총 50예 중 20예(40%)에서 배양검사와 REBA Fungus-ID®의 결과가 일치하였다. 20예 중 18예는 REBA와 배양검사에서 모두 T. rubrum이 동정되었고 나머지 2예는 REBA에서는 pan-fungus, 배양검사에서는 각각 Cladosporium spp., R. mucilaginosa로 진단되었으나, 본 실험에 사용된 얇은 막에는 Cladosporium spp.와 R. mucilaginosa에 대한 특이 probe가 없었으므로, 부착된 probe 이외의 진균감염을 똑같이 동정한 것으로 간주하여 일치한다고 판독하였다.
일치하지 않았던 30예(60%) 중 REBA FungusID®로 T. rubrum으로 판독된 20예에서 진균배양은 음성 소견을 보였으며, REBA Fungus-ID®에서 T. rubrum으로 진단된 다른 5예에서는 진균배양에서 Cladosporium spp. 1예, L. soppitti 1예, P. cvjetkovicii 1예, R. mucilaginosa 1예, 비피부사상균성사상균(non-dermatophytic mold) 1예가 동정되었다. REBA Fungus-ID®에서 T. mentagrophytes로 진단된 1예에서 진균배양은 음성 소견이었고, REBA FungusID®에서 Trichophyton spp.로 진단된 4예 중 1예는 진균배양 검사 음성, 나머지 3예는 각각 Cladosporium spp., C. europaea, R. mucilaginosa가 배양되었다.
고 찰
KOH 검사, 육안적 관찰과 같은 고식적 진단방법은 손발톱진균증에서 진균을 확인하기 위하여 널리 사용돼 왔다. KOH 검사는 KOH 전 처치 후 현미경으로 직접 관찰하는 것이고 육안적 관찰은 배양된 진균 분리주의 표현형 또는 생화학적 특성을 발견해내는 것이다[15]. 하지만 서론에서 언급하였듯이 이러한 방법은 진균을 빠르고 정확하게 진단하는 것을 방해하는 여러 가지 한계가 있다. 고식적 방법들은 낮은 민감도와 특이도를 가지고 또한 배양되고 검사자에 의해 판독이 내려지기까지 오랜 시간이 소요된다.
이러한 한계를 극복하기 위해서 앞서 언급된 많은 분자생물학적 방법이 소개되었다. 균종 특이 PCR은 한 가지 종만 검출할 수 있으며, multiplex PCR은 여러 균종을 동시에 검출하므로 신속하고 간편하지만 교차반응의 가능성이 있고, primer의 설계가 힘들다는 단점이 있다. 직접 염기서열분석법은 균종의 정확한 정보를 알려주지만 혼합감염 시 사용할 수 없다. RFLP 분석은 표준 균종과 비교해야 하므로 많은 시간과 노력을 필요로 하기 때문에 일반적인 진단 목적으로는 부적절하다[16]. ELISA는 민감도와 특이도가 매우 높은 우수한 진단법이나 비싼 장비가 필요하여 최근에는 임상적으로 사용이 줄어들고 있다[9],[17]. Real-time PCR은 높은 민감도와 특이도를 가졌지만 동시에 균을 검출하기 위해서는 고가의 장비와 숙련된 기술이 필요하다[18],[19].
PCR-REBA는 종 특이 ITS 서열에 기반한 PCRreverse line blot의 일종이다[11]. 얇은 막에 고정시킨 여러 개의 probe에 PCR 산물을 반응시켜 동시에 여러 개의 균주를 검출할 수 있다[20],[21]. 지금까지 PCR-REBA는 사람유두종 바이러스, 그리고 패혈증의 원인균을 확인하기 위해 사용돼 왔다[20]. 얇은 막에 포함시킬 수 있는 면적에 한계가 있어 많은 수의 probe를 모두 사용하지는 못하지만 특정한 감염증을 일으키는 균의 종류가 많지 않은 경우, PCR-REBA에 이러한 probe를 선별하여 포함시킬 수 있기 때문에 임상에서 사용이 유용하다는 장점이 있다.
손발톱진균증 진단용 kit인 REBA Fungus-ID®는 PCR-REBA의 원리를 기반으로 하며, 손발톱진균증의 흔한 원인균을 찾아낼 수 있도록 제작되었다. 한국에서 흔한 6종류의 피부사상균을 동정하는 실험이 있었고, 1 fg의 피부사상균 DNA만으로도 해당 진균을 정확하게 동정해내었다[12]. 또한 임상 분리주에서도 피부사상균을 포함하여 Candida 또는 Aspergillus spp.을 정확히 동정하였다[22].
이번 실험의 목적은 복합감염이 흔한 손발톱진균증에서 REBA Fungus-ID®과 고식적 진단방법인 진균배양의 진단 효율성을 비교하는 것이었다. REBA Fungus-ID®는 민감도에서 100%를 보여 56%를 나타낸 진균배양 검사보다 우수하였다. Yoo 등[23]의 2001년에서 2010년 사이에 발생한 손발톱진균증을 분석한 자료에 따르면 원인균종이 밝혀진 발톱진균증 총 338예에서 피부사상균이 80.5% (272/338), 효모균이 10.6% (36/338), 비피부사상균성사상균이 8.9% (30/38)였다. 피부사상균 중에서는 94.5% (257/272)가 T. rubrum, 5.5% (15/ 272)가 T. mentagrophytes로 나타났다. 이는 T. rubrum이 89.6% (43/48), T. mentagrophyte가 2.1% (1/48)로 동정된 본 실험 결과와 대체적으로 유사하였다. 균종이 밝혀진 338예 중 약 97%인 328예는 REBA Fungus-ID®에 사용된 probe로 동정 가능한 T. rubrum, T. mentagrophytes, C. albicans, C. parasilosis, C. tropicalis, A. niger, A. fumigatus, A. flavus, Scopulariopsis brevicaulis이었다.
REBA Fungus-ID®에서 T. rubrum, T. mentagro- phytes, Trichophyton spp.가 동정된 예 중에서 진균배양 검사에서는 다른 균이 동정된 예들이 8예가 있었다. 일반적으로 피부사상균의 병원성이 더 강하므로 Trichophyton spp.가 원인균이고, 진균배양 검사에서 동정된 균들은 오염 내지 상재균으로 생각하는 것이 합당하다.
REBA Fungus-ID®에서 pan-fungus로 동정된 2예는 배양검사에서 Cladosporium spp.와 R. mucila- ginosa가 각각 배양되었다. REBA Fungus-ID®의 pan-fungus가 배양검사와 일치하는지 알기 위하여 발톱에서 직접 추출된 DNA로 직접염기서열분석을 시도하였으나, 혼합감염으로 염기서열분석이 불가하였다. Cladosporium spp.와 R. mucilaginosa 특이 PCR을 시행하지는 못하였다. 추가적인 역학 연구 및 실험을 통해 흔히 관찰되는 다른 균종이 있다면, 그 균종에 대한 probe를 더 추가하는 것도 고려해보아야 할 것이다.
REBA Fungus-ID®는 결과 판독에서 발색반응의 강도 차이가 보이는 경우가 있었다(Fig. 3). 이러한 발색반응의 강도 차이는 복합감염이 있는 경우 probe에 없는 진균 DNA가 PCR 시 많이 증폭되어 상대적으로 적은 양의 probe 특이 진균 DNA의 증폭이 적게 일어났다고 생각한다. 발색반응의 강도가 어느 정도 이상을 보일 때 양성으로 판단할지에 대한 기준이 없어서 본 연구자는 각 막에 존재하는 색 대조군과 비교를 하여 비슷한 정도 이상의 발색반응이 일어나는 경우를 양성으로 판단하였다. 기준이 모호한 경우 판독자에 따라 결과 해석의 차이가 발생할 수 있으므로, 앞으로 이러한 기준에 대한 명확한 제시가 필요할 것으로 생각한다. 검사 전 과정은 사람의 손이 필요하다. 이러한 문제를 해결하기 위한 자동화를 해야 한다.
몇 가지의 보완이 이루어진다면 REBA FungusID®의 사용은 임상에서 피부과 의사가 손발톱진균증을 진단하고 치료하는 데에 많은 도움을 줄 것으로 생각되며, 앞으로 더 많은 대상자를 상대로 한 추가적인 연구가 필요할 것으로 생각한다.
결 론
손발톱진균증의 원인균을 검출하기 위해서 우리는 감수성이 낮은 진균배양 또는 복잡한 분자생물학적 방법을 활용한 방법을 사용하였다. PCRREBA는 민감도와 특이도가 높고 검체를 직접사용 가능하며 다른 분자생물학적 방법에 비해 상대적으로 가격이 싸서 손발톱진균증 진단에 유용하다. 본 연구자는 진균배양 검사와 PCR-REBA의 일종인 REBA Fungus-ID®의 진단 효용성을 비교하기 위해 영남대학교 병원에 내원한 환자 중 임상적으로 손발톱진균증이 의심이 되고 KOH 검사에서 양성 소견을 보이는 50명의 환자에서 50개의 검체를 채취하였고 각각의 검체에서 진균배양 검사 및 REBA Fungus-ID®를 시행하였다.
진균배양 검사와 REBA Fungus-ID®의 검사 민감도는 각각 56%와 100%였다. REBA Fungus-ID®에서 50예 중 43예에서 T. rubrum이 동정되었고, 4예는 Trichophyton spp., 1예는 T. mentagrophytes, 2예는 pan-fungus가 동정되었다. 진균배양 검사는 50예 중 28예에서 배양 소견을 보였다. 배양된 28예 중에서 18예는 T. rubrum이었고, R. mucila- ginosa와 Cladosporium spp.가 각 3예, C. europae, P. cvjetkovicii, L. soppittii, non-dermatophytic mold가 각각 1예씩 동정되었다. REBA Fungus-ID®와 진균배양 검사는 40%인 20예에서 일치 소견을 보였고, 16%인 8예는 결과가 상이하였으나 REBA Fungus-ID®에서는 피부사상균이 동정되고 진균배양 검사에서는 비피부사상균성진균이 동정되어 오염균으로 판정하였다. 44%인 22예는 REBA Fungus-ID®에서는 양성이었지만 진균배양 검사에서는 음성 소견을 보였다.
이와 같은 결과를 종합하여 볼 때 REBA Fungus -ID®는 임상 시료에서 직접 원인균을 동정할 수 있으며, 진단 민감도가 높고, 중복감염을 동정할 수 있는 손발톱진균증 진단 도구로 유용하다.
Conflict of interest
In relation to this article, I declare that there is no conflict of interest.
References
1. Chabasse D. Can we evaluate the frequency of onychomycosis? Ann Dermatol Venereol 2003;130:1222 -1230
Crossref
Google Scholar
2. Sigurgeirsson B, Baran R. The prevalence of onychomycosis in the global population - A literature study. J Eur Acad Dermatol Venereol 2014;28:1480-1491
Crossref
Google Scholar
3. Monad M, Lurati M, Baudraz-Rosselet F. Diagnosis of non dermatophyte onychomycosis and its relevance for treatment. Rev Med Suisse 2013;9:730-733
4. Weinberg JM, Koestenblatt EK, Tutrone WD, Tishler HR, Najarian L. Comparison of diagnostic methods in the evaluation of onychomycosis. J Am Acad Dermatol 2003;49:193-197
Crossref
Google Scholar
5. Gupta AK, Simpson FC. Diagnosing onychomycosis. Clin Dermatol 2013;31:540-543
Crossref
6. Jin HW, Kim HJ, Kim SH, Choi YI, Bang HE, Park SJ, et al. Evaluation of a PCR-reverse blot hybridization assay to identify six dermatophyte predominant in republic of Korea. Biomedical Science Letters 2014; 20:139-146
Crossref
Google Scholar
7. Bergmans AM, van der Ent M, Klaassen A, Bohm N, Andriesse GI, Wintermans RG. Evaluation of a singletube real-time PCR for detection and identification of 11 dermatophyte species in clinical material. Clin Microbiol Infect 2010;16:704-710
8. Khot PD, Fredricks DN. PCR-based diagnosis of human fungal infections. Expert Rev Anti Infect Ther 2009;7:1201-1221
Crossref
Google Scholar
9. Petinataud D, Berger S, Contet-Audonneau N, Machouart M. Molecular diagnosis of onychomycosis. J Mycol Med 2014;24:287-295
Crossref
10. Zeng X, Kong F, Halliday C, Chen S, Lau A, Playford G, et al. Reverse line blot hybridization assay for identification of medically important fungi from culture and clinical specimens. J Clin Microbiol 2007; 45:2872-2880
Crossref
Google Scholar
11. Bregmans AM, Schouls LM, van der Ent M, Klaassen A, Bohm N, Wintermans RG. Validation of PCRreverse line blot, a method for rapid detection and identification of nine dermatophyte species in nail, skin and hair samples. Clin Microbiol Infect 2008; 14:778-788
Google Scholar
12. Kim H, Jin H, Kim S, Wang HY, Choi Y, Bang H, et al. PCR-reverse blot hybridization assay for species identification of dermatophytes. Korean J Med Mycol 2011;16:86-89
Google Scholar
13. Bang H, Park S, Hwang J, Jin H, Cho E, Kim DY, et al. Improved rapid molecular diagnosis of multidrugresistant tuberculosis using a new reverse hybridization assay, REBA MTR-MDR: J Med Microbiol 2011;60: 1447-1454
Google Scholar
14. Kim S, Lee D, Park S, Kim TU, Jeon BY, Park KH, et al. REBA HPV-ID(R) for efficient genotyping of human papillomavirus in clinical samples from Korean patients. J Med Virol 2012;84:1248-1253
Crossref
Google Scholar
15. Weitsman I, Summerbell RC. The dermatophytes. Clin Microbiol Rev 1995;8:240-259
Crossref
16. Dhip I, Fathallah A, Yaacoub A, Hadj Slama F, Said MB, Zemni R. Multiplex PCR assay for the detection of common dermatophyte nail infections. Mycoses 2014;57:19-26
Google Scholar
17. Verrier J, Monad M. Diagnosis of dermatophytosis using molecular biology. Mycopathologia 2017;182: 193-202
Crossref
18. Hui D, Xue-cheng S, Ai-e X. Evaluation of reflectance confocal microscopy in dermatophytosis. Mycoses 2013;56:130-133
Crossref
Google Scholar
19. Idriss MH, Khalil A, Elston D. The diagnostic value of fungal fluorescence in onychomycosis. J Cutan Pathol 2013;40:385-390
Crossref
Google Scholar
20. Choi Y, Wang HY, Lee G, Park SD, Jeon BY, Uh Y, et al. PCR-reverse blot hybridization assay for screening and identification of pathogens in sepsis. J Clin Microbiol 2013;51:1451-1457
Crossref
Google Scholar
21. Kim JY, Choe YB, Ahn KJ, Lee YW. Identification of dermatophytes using multiplex polymerase chain reaction. Ann Dermatol 2011;23:304-312
Crossref
Google Scholar
22. Park SY, Kim BK, Wang HY, Kim SH, Kim HJ, Lee HY, et al. PCR-reverse blot hybridization assay for fast and accurate identification of causative species in superficial fungal infections. Clin Exp Dermatol 2016;41:359-365
Crossref
Google Scholar
23. Yoo JY, Suh MK, Ha GY, Kim HS. A clinical and mycological study of onychomycosis in the elderly over 10 years (2001-2010). Korean J Med Mycol 2014;19:83-92
Google Scholar